PD Dr. Volker Huß
PD Dr. Volker Huß
Forschungsprojekte
Molekulare Systematik von Grünalgen
Die klassische Taxonomie von Grünalgen anhand von morphologischen Merkmalen hat sich vor allem bei einzelligen Formen vielfach als problematisch erwiesen. Wir untersuchen daher die natürliche phylogenetische Verwandtschaft solcher Organismen mit Hilfe verschiedener molekularer Methoden. Neben „klassischen“ Methoden wie der Bestimmung der DNA-Basenzusammensetzung und quantitativer DNA/DNA Hybridisierung zur Artbestimmung anhand von DNA-Reassoziationskinetiken, steht die vergleichende Sequenzanalyse der stark konservierten nukleären ribosomalen 18S DNA sowie der plastidären ribosomalen 16S DNA für die Einordnung der Organismen in einen globalen Stammbaum im Vordergrund. Zur Feinauflösung nahe verwandter Gattungen und Arten, bzw. zur intraspezifischen Unterscheidung verschiedener Isolate verwenden wir die weitaus variablere Internal-Transcribed-Spacer (ITS) Region der ribosomalen RNA Gene. Von allen erhaltenen ribosomalen RNA Sequenzen erstellen wir Sekundärstrukturmodelle, die sowohl als Hilfestellung für das Sequenz-Alignment dienen als auch der Charakterisierung ungewöhnlicher Insertionen wie Gruppe I Introns oder in einem Fall eines „Double Hairpin Elementes“. Phylogenetische Stammbäume werden mit gängigen Methoden wie Neighbor Joining, Maximum Parsimony und Maximum Likelihood berechnet, die im Programmpaket „PAUP“ implementiert sind. Im Mittelpunkt unserer Untersuchungen steht die Gattung Chlorella und verwandte Algen wie die chlorophyllfreie, heterotrophe Gattung Prototheca, die tier- und menschenpathogene Arten enthält.
Grünalgen als Symbionten
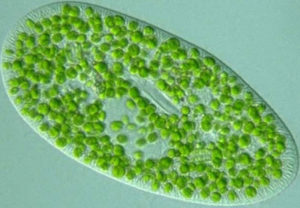
Verschiedene einzellige Grünalgen kommen als intrazelluläre Symbionten in unterschiedlichen Organismengruppen vor. Hauptsächlich handelt es sich dabei um Chlorella oder Chlorella-ähnliche Algen, die zum Teil obligate Symbiosen mit beispielsweise Nesseltieren wie Hydra oder Ciliaten wie Paramecium (Abb. 1) eingehen. Eine völlig neuartige, erst kürzlich entdeckte Symbiose stellt das Vorkommen intrazellulärer Vorstufen einer einzelligen Grünalge der Gattung Coccomyxa in Ginkgo-Zellen dar. Es handelt sich hierbei um den ersten Fall einer mutualistischen Symbiose zweier phototropher Organismen. Da manche der hier aufgeführten Symbionten bisher nicht kultiviert werden können, stellen die oben beschriebenen Methoden der molekularen Systematik die einzige Möglichkeit einer exakten taxonomischen Zuordnung dar.
Biodiversität extrem saurer Biotope

Mit Hilfe der „Environmental PCR“ Technik läßt sich die Biodiversität spezieller Biotope ermitteln. Dazu wird aus Proben eines Biotops die Gesamt-DNA isoliert und die darin enthaltenen verschiedenen ribosomalen RNA-Gene mit universellen PCR-Primern amplifiziert. Nach Klonierung und Sequenzierung der einzelnen 18S rRNA-Gene, können die im Biotop erhaltenen Organismen in einen phylogenetischen Stammbaum eingeordnet werden ohne dass dabei eine Isolierung und Kultivierung der Organismen erforderlich ist. Wir untersuchen mit dieser Methode die Biodiversität einzelliger Eukaryonten in zwei extrem sauren Tagebaurestseen in der Lausitz (Sachsen; Abb. 2) und der Oberpfalz mit pH-Werten von 2,3 und 2,7. Die Ergebnisse zeigen, dass viele der dort vorkommenden Organismen nicht einmal auf Ordnungs- oder Klassenniveau beschriebenen Taxa zugeordnet werden können. In einem Fall läßt sich die 18S rDNA-Sequenz sogar keiner der bisher bekannten Evolutionslinien der Eukaryonten zuordnen.
